2020年诺贝尔化学奖授予了Emmanuelle Charpentier和Jennifer A. Doudna,以表彰她们发展了基于CRIPSR-Cas9系统的基因编辑工具。但CRISPR-Cas9系统在进行基因编辑的过程中有可能会脱靶产生DNA双键断裂(double strand break, DSB),从而造成基因组的不稳定【1】。
Broad institute的David R. Liu教授在2016年在CRISPR-Cas9系统的基础上通过融合脱氨酶(rAPOBEC)和尿嘧啶糖基化酶抑制剂(UGI)发展了不会产生DNA双键断裂就可以进行基因组特定位点的C-to-T编辑的胞嘧啶碱基编辑器(cytosine base editor, CBE)【2】。由于CBE系统在编辑过程中不产生DNA双链断裂,所以一般认为CBE系统比CRISPR-Cas9系统拥有更高的安全性。但是,在真正将CBE应用到临床进行遗传疾病治疗前,有必要对其安全性与特异性进行评估。
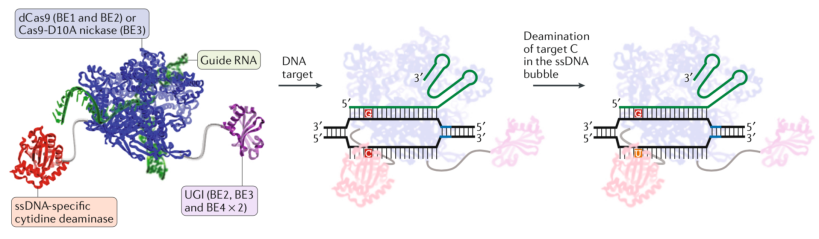
图- 1 CBE通过产生中间产物脱氧尿嘧啶(dU),实现碱基编辑【3】
针对CBE造成的DNA脱靶(off-targets)效应,目前已有的全基因组检测技术包括:(1)基于体外生化孵育后测序的Digenome-seq技术【4】;(2)基于检测Cas9蛋白介导产生DSB的GUIDE-seq【5】,CIRCLE-seq【6】等技术;(3)基于生物信息学序列预测的Cas-OFFinder【7】等软件;(4)基于单细胞、单克隆技术扩增后进行全基因组测序的技术【8-10】等。然而,依然缺乏灵敏、高效的检测手段,无偏向性地鉴定CBE在体内造成的脱靶位点。
2021年6月7日,北大-清华生命科学联合中心、北京大学生命科学学院伊成器课题组在Nature Methods杂志发表了题为“Detect-seq reveals out-of-protospacer editing and target-strand editing by cytosine base editors”的文章。研究人员巧妙地利用了CBE在进行靶向编辑过程中会产生中间产物脱氧尿嘧啶(dU)这一特点,对CBE产生的dU进行生物素(biotin)标记、富集;同时在dU位点的附近,将正常胞嘧啶C替换为d5fC,结合后续的化学标记反应【11-13】,产生串联C-to-T的信号从而增敏脱靶编辑的检测,开发了名为Detect-seq(dU-detection enabled by C to T transition during sequencing)的CBE脱靶检测技术。


图- 2 Detect-seq实验流程示意图
研究人员首先在人胚肾细胞HEK293T和人乳腺癌细胞MCF-7转染BE4max,分别使用4种、2种不同的sgRNA对基因组靶向位点进行C-to-T的编辑;同时对这些样本中进行了Detect-seq检测。通过Detect-seq检测发现,除RNF2位点外,其它靶向位点均可以产生几十至数百个Cas9依赖型的脱靶位点。

图- 3 在三种不同sgRNA样品中鉴定到的Cas依赖型的脱靶位点。其中蓝色圆圈代表脱靶位点(off-targets),红色方块代表靶向位点(on-target) EMX1 n=48,VEGFA_site_2 n=511, HEK293_site_4 n=245
随后,研究人员比较了由Detect-seq鉴定到的脱靶位点与之前研究报道的使用体外生化孵育Digenome-seq方法或基于Cas-OFFinder软件预测的脱靶位点。通过靶向扩增子测序的验证(targeted amplicon sequencing),由Detect-seq鉴定的脱靶位点均可以在验证实验中表现出显著高于背景的C-to-T的突变;而仅仅被体外生化孵育Digenome-seq方法或基于Cas-OFFinder软件预测到的脱靶位点,则没有表现出显著高于测序背景的C-to-T的突变。进一步的生物信息学分析则表明,Detect-seq鉴定到的脱靶位点显著富集于活跃转录的基因区、基因启动子区等开放染色质区域,与体外实验和计算机预测结果呈现了鲜明的对比。
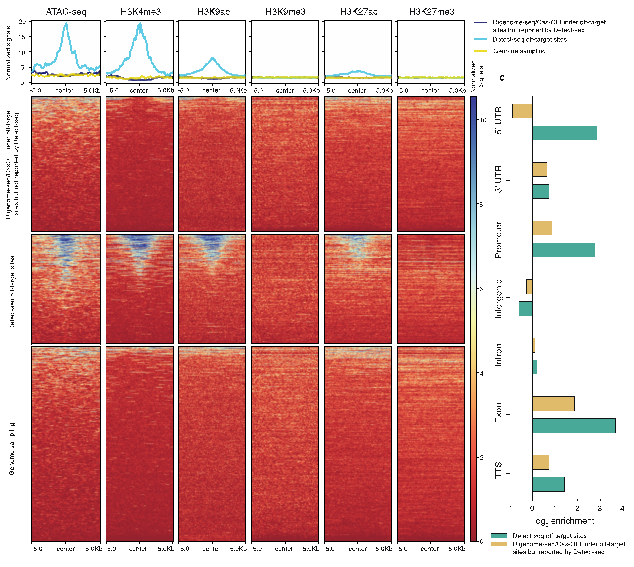
图- 4 Detect-seq技术与体外孵育检测技术Digenome-seq及预测软件Cas-OFFinder相比,鉴定到的位点富集于活跃转录区等染色质开放区域。
在Detect-seq之前,领域内常使用检测DNA双链断裂(DNA double strand breaks, DSBs)的技术(如GUIDE-seq,CIRCLE-seq等)检测由Cas蛋白核酸内切酶切割DNA双链造成的脱靶效应。并以此来替代CBE系统造成的脱靶效应。在文章中,研究人员也比较了Detect-seq与GUIDE-seq在三种sgRNA样品中鉴定到的Cas依赖型脱靶位点。结果发现,除EMX1位点外,剩余两个sgRNA样品中Detect-seq均检测出远多于GUIDE-seq的Cas依赖型脱靶位点。这提示了Cas蛋白的结合活性造成的脱靶与其核酸内切酶造成的脱靶可能不同。
有趣的是,研究人员基于Detect-seq技术还发现了两种新型的Cas依赖型的脱靶编辑:sgRNA结合区域外编辑(out-of-protospacer edit)及靶向链编辑(target-strand edit)。
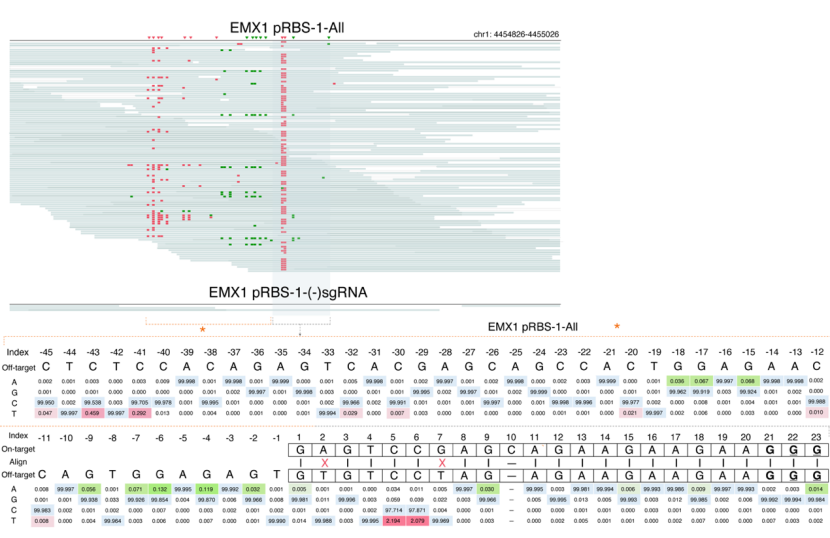
图- 5 CBE介导的sgRNA结合区域外编辑与靶向链编辑 sgRNA结合区域使用阴影标出,IGV图中红色方块代表正链C-to-T突变,绿色方块代表反链C-to-T突变;图下半部分为该区域靶向扩增子测序结果。
sgRNA结合区域外编辑即C-to-T的编辑发生在sgRNA结合区域外;而靶向链编辑则是C-to-T编辑发生在sgRNA与DNA结合链。根据Detect-seq的结果,上述两种编辑普遍存在于Cas依赖型的脱靶位点的结合区域。值得一提的是,虽然RNF2位点未发现Cas依赖型的脱靶,但是在其靶向位点的靶向链发现了显著高于测序背景的C-to-T编辑。根据后续的机器学习计算结果,研究者认为sgRNA 5’端错配和PAM附近的错配可能是影响sgRNA结合区域外编辑和靶向链编辑的关键因素。
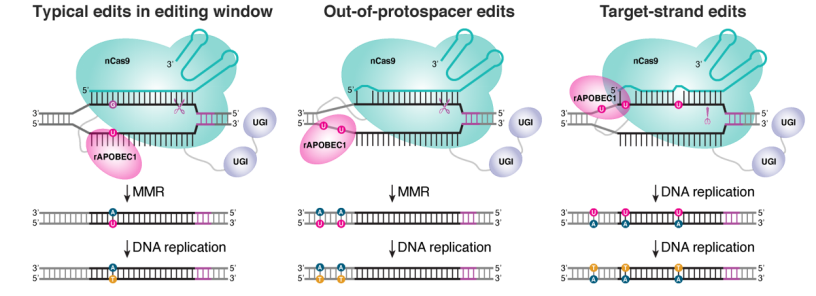
图- 6 sgRNA结合区域外编辑与靶向链编辑模式图
随后,研究人员发现,除了上述Cas依赖型的脱靶位点,Detect-seq技术也可以在转染BE4max的样品中检测到一些信号明显较弱、删除sgRNA后依然存在,但删除脱氨酶后降低至背景水平的Cas非依赖型脱靶。对于上述位点进行生物信息学分析后发现,该种位点主要发生在TC motif中的C中,与之前报道的rAPOBEC偏好性相同【14】。
Detect-seq技术为碱基编辑领域内提供了一种能够高灵敏、高特异、无偏好性地检测细胞内的CBE脱靶位点的方法。脱靶效应理解的加深,也将为阐明CBE的催化机制、优化改造更安全的CBE工具带来新的视角与可能。为了便于Detect-seq技术的使用,研究人员还为Detect-seq编写了配套的生物信息学分析软件(代码已上传至Github,详见论文Methods部分)。
原文链接:
https://doi.org/10.1038/s41592-021-01172-w
新闻稿出处:BioArt
参考文献
1.Jiang, F. and J.A. Doudna, CRISPR-Cas9 Structures and Mechanisms. Annu Rev Biophys, 2017. 46: p. 505-529.
2.Komor, A.C., et al., Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature, 2016. 533(7603): p. 420-4.
3.Rees, H.A. and D.R. Liu, Base editing: precision chemistry on the genome and transcriptome of living cells. Nat Rev Genet, 2018. 19(12): p. 770-788.
4.Kim, D., et al., Genome-wide target specificities of CRISPR RNA-guided programmable deaminases. Nat Biotechnol, 2017. 35(5): p. 475-480.
5.Tsai, S.Q., et al., GUIDE-seq enables genome-wide profiling of off-target cleavage by CRISPR-Cas nucleases. Nat Biotechnol, 2015. 33(2): p. 187-197.
6.Tsai, S.Q., et al., CIRCLE-seq: a highly sensitive in vitro screen for genome-wide CRISPR-Cas9 nuclease off-targets. Nat Methods, 2017. 14(6): p. 607-614.
7.Bae, S., J. Park, and J.S. Kim, Cas-OFFinder: a fast and versatile algorithm that searches for potential off-target sites of Cas9 RNA-guided endonucleases. Bioinformatics, 2014. 30(10): p. 1473-5.
8.Jin, S., et al., Cytosine, but not adenine, base editors induce genome-wide off-target mutations in rice. Science, 2019. 364(6437): p. 292-295.
9.McGrath, E., et al., Targeting specificity of APOBEC-based cytosine base editor in human iPSCs determined by whole genome sequencing. Nat Commun, 2019. 10(1): p. 5353.
10.Zuo, E., et al., Cytosine base editor generates substantial off-target single-nucleotide variants in mouse embryos. Science, 2019. 364(6437): p. 289-292.
11.Xia, B., et al., Bisulfite-free, base-resolution analysis of 5-formylcytosine at the genome scale. Nat Methods, 2015. 12(11): p. 1047-50.
12.Zhu, C., et al., Single-Cell 5-Formylcytosine Landscapes of Mammalian Early Embryos and ESCs at Single-Base Resolution. Cell Stem Cell, 2017. 20(5): p. 720-731 e5.
13.Zeng, H., et al., Bisulfite-Free, Nanoscale Analysis of 5-Hydroxymethylcytosine at Single Base Resolution. J Am Chem Soc, 2018. 140(41): p. 13190-13194.
14.Saraconi, G., et al., The RNA editing enzyme APOBEC1 induces somatic mutations and a compatible mutational signature is present in esophageal adenocarcinomas. Genome Biol, 2014. 15(7): p. 417.

